推荐产品
公司新闻/正文
重要!您的能量代谢数据是否进行了正确的归一化?
144 人阅读发布时间:2025-03-28 10:31
数据归一化的重要性
功能生物数据的归一化是原始数据展示和后续分析工作流程中的关键因素,以确保对结果的准确一致的解析。XF 代谢分析在这方面也是如此,大多数实验需要进行某种形式的归一化。无论是比较不同的细胞类型、基因修饰还是化合物处理,都必须根据共同的参数将数据归一化以便正确比较。XF 分析可在包括细胞数、基因组 DNA 和总细胞蛋白质的多个参数层面进行归一化。本文档重点介绍使用细胞数(或细胞数的替代参数)来归一化 XF 速率数据(OCR、ECAR、PER)的方法。
影响细胞密度和细胞增殖速率的因素
准备 XF 分析时,许多因素会影响细胞密度(每孔细胞数),包括:增殖速率、细胞分化程度、细胞死亡速率和点样速率或细胞粘附效率。增殖速率是关键的,因为大多数依赖贴壁的细胞在 XF 分析之前至少需要经过一次过夜培养,细胞数量在这一培养期间会发生变化。在引入干预措施(如基因修饰、慢性药物治疗等)时了解增殖速率尤其重要,因为这些措施通常会导致细胞生长速率发生变化,因此在分析和解析 XF 数据时必须考虑在内。通过绘制细胞数与时间关系图,可凭经验确定目标细胞类型的生长速率。理想策略是通过考虑实验组之间倍增时间的差异适当调整每孔接种细胞数;以便在 XF 分析时最/大程度减小组间细胞数的差异。通过测量每孔细胞数或细胞含量,可将不好控制的最终细胞计数的任何变化进行归一化。另一重要考虑因素是如果要在数天或数周时间内进行相似类型的 XF 分析,则需要保持细胞接种和 XF 分析之间的培养时间恒定。
归一化方法
A. 总细胞蛋白质
根据总细胞蛋白质进行归一化的过程相对较快且经济,采用几乎任意标准酶标仪即可实现。将细胞裂解,并通常将一部分孔内容物通过 Bradford 或 BCA 蛋白质检测试剂进行定量分析。建议始终包含标准蛋白质浓度曲线以确保准确定量分析,并允许不同分析之间数据的绝对比较。图1 显示了已根据总细胞蛋白质归一化的原始 OCR 和 ECAR 数据。该方法虽然简单易行,但其隐含的假设是,对细胞的任何干预措施都不会明显改变细胞总蛋白质含量。如果细胞处理导致线粒体生物合成发生变化,从而改变细胞的蛋白质含量,那么这种归一化方法就可能出现问题,可能掩盖真正的活性差异[1]。下文将详细介绍线粒体生物合成的评估。如果不同实验组之间的细胞外基质蛋白质含量存在显著差异,或如果培养板基底包被了含有蛋白质的细胞粘附剂(如胶原蛋白、层粘连蛋白、Matrigel®),则总蛋白质归一化处理也不适用。
B. 核 DNA
在总蛋白质或细胞计数可能不相关或不可行的情况下,每孔核 DNA 含量可用于归一化 XF 速率数据[2]。该方法基于以下假设:与上述不适合使用总细胞蛋白质的某些实例不同,核 DNA 含量与细胞数呈线性相关。掺入 dsDNA 的各种荧光或比 色染料通常用于定量分析核 DNA。参考文献 [3] 和 [4] 对这些方法和染料进行了全面综述,包括使用 PicoGreen 和 CyQuant 试剂的示例性数据。与总蛋白质分析一样,建议使用参比 dsDNA(如 λDNA)标准曲线以确保准确定量分析,并允许不 同数据组的绝对比较。
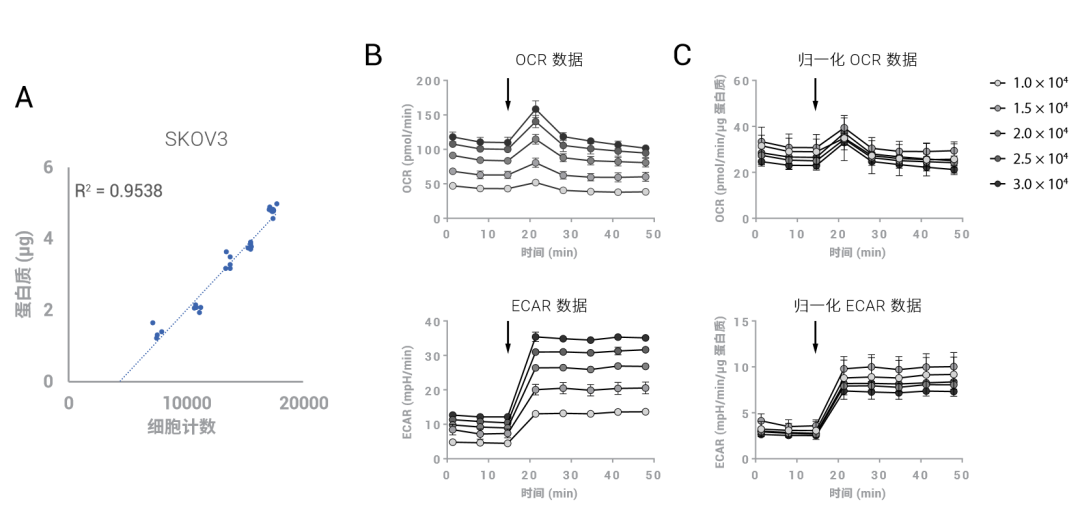
图 1. 使用 SKOV3 细胞的总细胞蛋白质进行 XF 数据归一化的示例。将细胞以 1 × 104、1.5 × 104、2 × 104、2.5 × 104、3 × 104 细胞/孔点样于 XF96 组织培养微孔板中 (n = 6),培养 24 小时,然后评估基础和应激 OCR 和 ECAR(由 1.0 µmol/L 寡霉素 + 0.5 µmol/L FCCP 诱导的应激条件,终浓度,箭头)。A) 使用 Cytation 1 计数的细胞数与总细胞蛋白质值呈线性相关。B) 不同点样密度下的基础和应激状态的原始 OCR 和 ECAR 值。C) 不同点样密度下的基础和应激状态的 OCR 和 ECAR 值(已根据总细胞蛋白质归一化处理)(平均值 ± SD,n = 6)
C. 细胞计数
针对 XF 代谢速率数据最稳定的归一化方法包括通过细胞直接 成像或染色细胞核成像,对微孔板的每孔细胞数进行计数。两种成像方法都需要专用的高通量自动成像仪器。许多成像系统可在 XF 分析后直接计数细胞,包括 BioTek Instruments 的 Cytation 1,该系统可用于直接细胞计数和核染色细胞计数。使用可渗透细胞的核染色剂进行成像及定量分析细胞数比直接细胞成像更有优势,因前者工作流程更简单(如不需要固定细胞)并可实现自动化,同时无需制备参比样品。因为细胞直接计数需要进行显微图像采集及后续图像分析,因此要求细胞分散良好且形态边界清晰(如 A549 或 SKOV3)。对于分散程度较低的细胞或具有聚集形态的细胞(如 MCF7),核染色方法更适用。此外,该方案的无损特性使其可与其他下游分析兼容,例如测量总蛋白质、PCR 或免疫染色。注意,可渗透细胞的核染料可通过 XF 探针板上的加药口原位直接加入到培养液中,如果所有四个加药口均在使用,可在 XF 分析后加入。图2 显示了使用 Cytation 1 进行原位核染色和计数,然后用于 XF 细胞能量代谢表型测试数据归一化(使用 SKOV3 细胞)的示例。图 3 显示了使用形态和核大小各不相同的三种不同细胞类型的进一步归一化示例。如需了解上述成像/归一化方法的详细信息(包括工作流程和比较示例),可访问官网以下应用简报:“使用 BioTek Cytation 5 通过原位细胞计数对安捷伦 Seahorse XF 数据进行归一 化”。
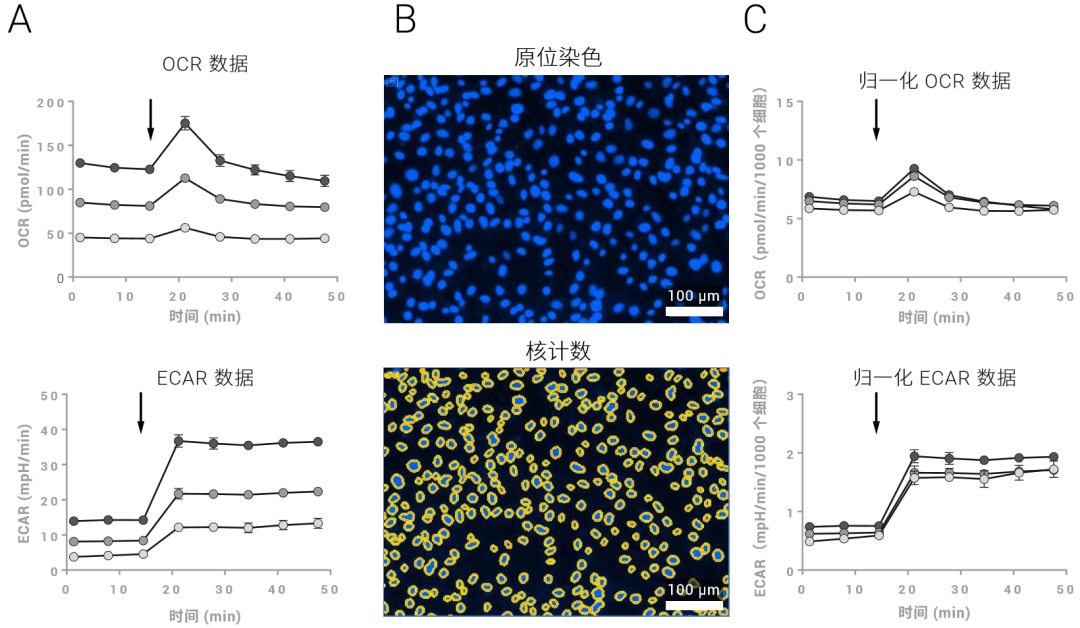
图 2. 使用原位核染色和原位细胞计数的 XF 数据归一化的示例。将 SKOV3 细胞以 1 × 104、2 × 104、3 × 104 细胞/孔进行点样,培养 24 小时,进行 XF 细胞能量代 谢表型测试,然后进行图像分析。A) 原始 OCR 和 ECAR 随寡霉素 + FCCP 加药(分别为 1.0 µmol/L 和 0.5 µmol/L 终浓度)而发生变化(箭头),包括 20 µmol/L Hoechst 33342(2 µmol/L 终浓度)。B) 由 Hoechst 33342 荧光标记的细胞核(上图)以及使用 Cytation 1 鉴定和分析的细胞核(下图)的代表性图像。C) OCR 和 ECAR 通过原位核染色细胞计数进行归一化(平均值 ± SD,n = 4)
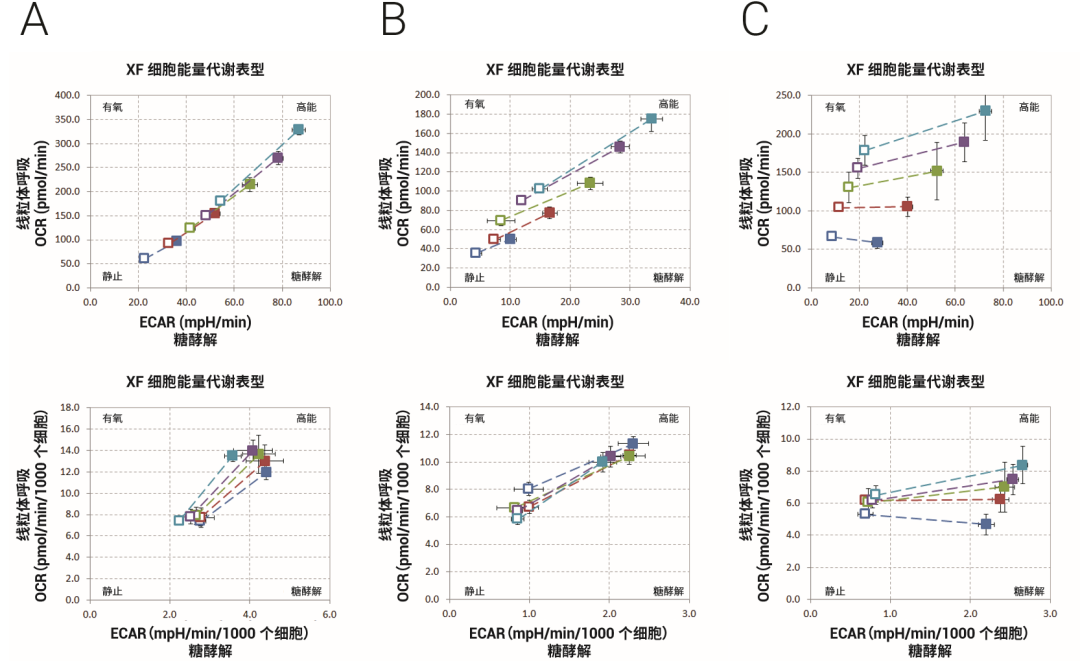
图 3. 通过 HT-29 (A)、MCF7 (B) 和 RAW264.7 (C) 原位核染色使用 Cytation 1 进行 XF 数据归一化的示例。对于 HT-29 和 MCF7,将细胞以 1 × 104、1.5 × 104、 2 × 104、2.5 × 104、3 × 104 细胞/孔,对于RAW264.7,将细胞以 1.5 × 104、2.8 × 104、3.0 × 104、3.8 × 104、4.5 × 104 细胞/孔接种于 XF96 组织培养微孔板中,培养 24 小时通过寡霉素 + FCCP 加药(分别为 1.0 µmol/L 和 0.5 µmol/L 终浓度),包括 20 µmol/L Hoechst 33342(2.0 µmol/L 终浓度),执行细胞能量代谢表型测试。由 Seahorse XF 细胞能量代谢表型报告生成器生成的 XF 能量图在归一化前(上图)后(下图)的比较(平均值 ± SD,n = 6)
特殊情况:
非增殖细胞:包括培养一段时间但不复制的原代和/或有丝分裂后细胞(例如皮层神经元、新生大鼠心室肌细胞、棕色脂肪细胞、分化的 iPSC 等[5, 6])。通常会在接种到 XF 组织培养微孔板之前计数细胞,获得初始值。然而,仍然建议在 XF 分析后执行一些相关的归一化方法,将培养过程中分离或丧失活性可能造成的任何细胞损失考虑在内。
强制粘附细胞:一些细胞或 XF 应用需要将细胞强制粘附(通常采用离心)到 XF 组织培养微孔板上(例如 T 细胞活化分析)。在这些情况下,通常在分析前进行定量细胞计数,并将已知数量的细胞引入每个孔中。同样,在分析后对孔内活细胞数进行评估仍然有用,将分析期间的分离可能造成的任何细胞损失考虑在内。
三维样品(例如细胞球体)可以根据样品大小或体积进行归一化。细胞球体通常在单独的容器中生长,从数百个到数千个细胞开始。虽然更难以通过总蛋白质、核 DNA 或细胞计数来评估,但使用细胞球体直径、总球体体积等几何参数可进行计算并用作归一化参数[7] 。
分离的线粒体或突触体:在 XF 仪器中使用分离的线粒体或突触体需要在 XF 分析前定量分析样品蛋白质含量,并接种优化的量。在这些情况下,通常不需要在分析后对线粒体或突触体蛋白进行评估,因此也不需要进行归一化[8, 9]。
选择相关性最高的归一化方法
归一化方法的初始选择通常以所分析的样品类型开始。以下方案介绍了选择最佳方法的决策过程。本文介绍的归一化技术各自具有各自的优缺点,没有单个归一化方法普遍适用于每种实验设计及后续分析。
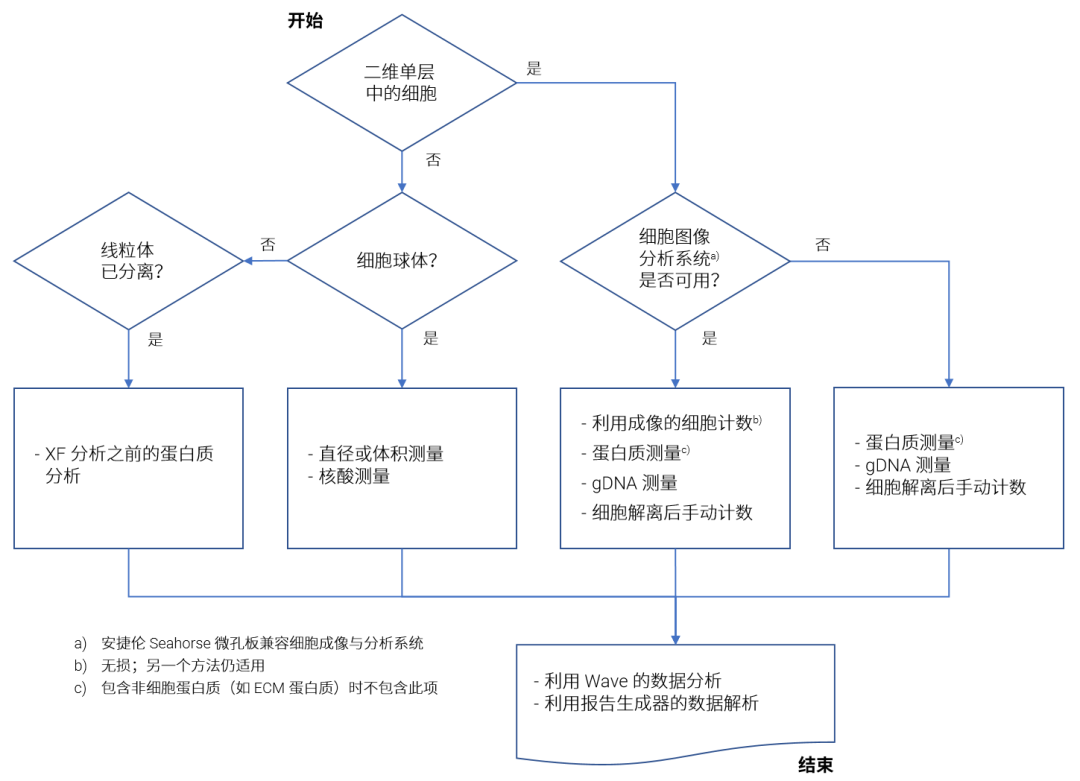
在多数情况下,可以应用多种归一化方法。然而,不推荐任何基于细胞代谢(例如 MTT 分析、总 ATP 水平)定量分析细胞数的方法,因为 XF 分析专门设计用于测量细胞代谢,因此需要应用与代谢功能无关的正交归一化技术。下表 1 列出了上述归一化方法的主要优点和缺点:
|
归一化方法 |
优点 |
缺点 |
|
总细胞蛋白质 |
- 价格低 - 与大多数酶标仪兼容 |
-样品传输可能会带来误差 -与 ECM 包被板不兼容 -需破坏细胞来获得蛋白质 -如果线粒体生物合成发生变化则不适用 |
|
基因组 DNA |
- 与大多数荧光酶标仪兼容 |
- 样品传输可能会带来误差 - 不适用于多核细胞 - 需破坏细胞来获得核 DNA |
|
细胞成像 |
-获得细胞和/或细胞核计数的最直接方法 -XF 分析后无需处理 -细胞仍可用于下游应用 -可与 ECM 包被板兼容 |
-需要专门的细胞计数仪器 |
表1 注意,所使用的所有归一化方法的核心是假设细胞数和被测信号之间存在线性关系;每个细胞的分析物含量保持不变。但是,这种假设并不总是有效。例如,通过线粒体生物合成而增加代谢活性的细胞,每个细胞都具有更高的 OCR,但是,如果采用总细胞蛋白质归一化方法,则这种呼吸差异可能被低估甚至掩盖。如上文所述,如果疑似出现线粒体生物合成,则总细胞蛋白质不适用于 XF 分析数据归一化,而应采用基因组DNA,或最好采用细胞数。
细胞数与细胞活性
归一化 XF 数据时要考虑的另一重要因素是细胞数与细胞活性之间的关系,即每孔或处理组中有多少细胞具有活性?细胞增殖和/或细胞毒性的正交测量与 XF 数据结合使用时,这一点尤其重要。如果需要测量细胞活性,则使用不受任何 XF 分析试剂盒试剂强处理影响的方法至关重要,因为这些试剂会抑制线粒体和/或糖酵解功能。需要注意的是,其中包括依赖于细胞 NAD(P)H 氧化还原酶的活性分析,例如 MTT 和 MTS 分析。如果将测量总细胞 ATP 水平作为细胞活力/增殖的代表,也应谨慎,因为近期研究表明,将细胞 ATP(和 MTT)与绝对细胞数量相关联时存在差异[10]。其他活性分析(包括 MultiToxFluor 细胞毒性分析)与 XF 分析试剂兼容,并可以在 XF 分析后使用以获得活细胞与死细胞的比率。注意,细胞活性通常以相对比或百分比表示,因此必须测量细胞的绝对数量以准确归一化 XF 数据。
归一化的其他注意事项
如上所述,有些情况不应将某一归一化方法应用于 XF 数据。这些情况通常与每个细胞的线粒体数量/质量的变化(即线粒体生物合成与线粒体自噬)、mtDNA 编码蛋白质表达变化和/或线粒体电子传递与氧化磷酸化复合物(甚至复杂亚基)相互之间的化学计量有关。
在上述情况下,细胞总蛋白质不应用于归一化,因为细胞生物学的重要差异可能被掩盖。细胞计数和/或 gDNA 适用于上述情况。如果疑似线粒体数量/质量发生变化,则通过 qRTPCR 测量 mtDNA 或 mtDNA:nDNA 比的相对变化是适用的正交验证方法[1, 11]。在上述线粒体质量/数量变化的情况下,建议设置线粒体生物合成的阳性对照(例如用 AICAR、二甲双胍等处理 细胞[12]),以确定细胞和线粒体响应的动态范围和灵敏度。检测 ETC/OxPhos 复合物的相对含量或化学计量变化,可通过将多种电子传递链蛋白的免疫印迹根据一种或多种细胞质蛋白进行标准化来评估[13, 14]。
在 Wave 中“应用归一化”并使用“基线”按钮:
用于查看 XF 数据的 Wave 软件具有内置的“基线”功能,可将绝对 XF 速率数据转换为相对 (%) 比例。通常,基线设定为首次进样前的速率。如需尽量减小由于细胞接种或增殖变化引起的差异,基线数据是最合适的,且有助于显示强处理/加药导致的速率化。
Wave 软件中的“归一化”功能提供了一种简单方法,将归一化数据应用于测量的速率数据(OCR、ECAR、PER)。为了使用归一化功能,如上文所述,需要对板孔进行细胞数、蛋白质浓度、DNA 含量的独立评估。
要在 Wave 中归一化数据,需使用以下三个组成部分:
归一化值(必需):由孔独立评估获得的数值数据(细胞计数、蛋白质浓度、DNA 含量)
归一化单位(必需):此字母数字字段描述了用于数据归一化的参数单位。包括归一化值的度量单位(例如“细胞”,“mg”,“ ng”等)
归一化缩放因子:此数字确定比率数据将要缩放到的值。
默认值为 1,可进行调整
然而,这一功能不应看作是归一化的替代,因为关键信息可能在转换时丢失(图4)。应考虑数据展示和比较不同实验室之间结果的能力,因此建议报告绝对归一化值。因此,基线功能应仅用于在分析开始时具有完全相同条件的组之间的初始比较,并且应采用相关的绝对归一化方法。
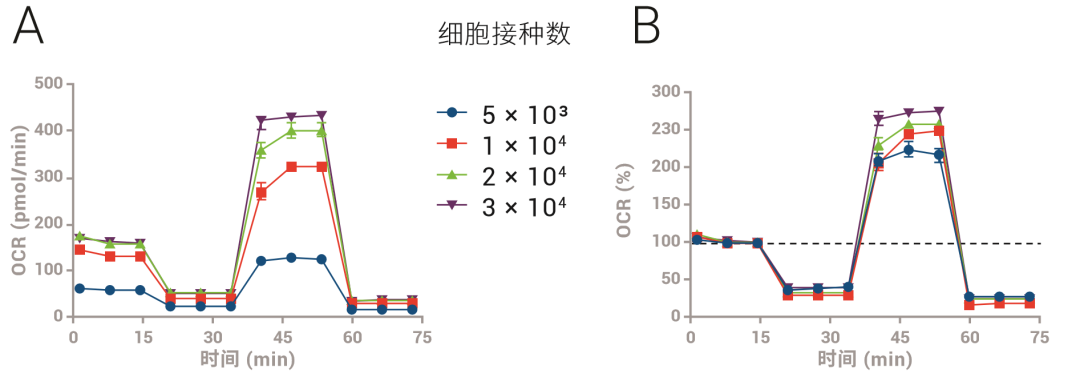
图 4. 绝对与基线 OCR 数据:图 A 显示绝对 OCR,其呼吸速率与细胞数量相关。图 B 显示,在每个细胞密度下,对 XF 细胞压力测试化合物的响应大致相等,但是采 用基线功能转换时,丢失了 OCR 差异相对于接种密度的信息。
归一化仪器推荐
Cytation 活细胞成像系统与Seahorse XF分析仪整合,能够形成一整套操作简便的完美细胞代谢成像归一化系统。

由于Cytation系列产品优秀的双平台设计,整合了基于PMT检测的多功能微孔板检测系统,研究者通过该系统的吸收光、荧光、发光检测模块,可以开展基于总蛋白、总DNA的细胞归一化检测;而Cytation配备的自动化成像模块,能够开展基于细胞计数的数据标准化,通过此方法,可对比不同孔、细胞板甚至是不同实验批次的数据,从而得到更具生物学意义的实验结果。
参考文献:
1. Liu, T.F., et al., Sequential actions of SIRT1-RELB-SIRT3 coordinate nuclear-mitochondrial communication during immunometabolic adaptation to acute inflammation and sepsis. J Biol Chem, 2015. 290(1): p. 396-408
2. Lorenz, C., et al., Human iPSC-Derived Neural Progenitors Are an Effective Drug Discovery Model for Neurological mtDNA Disorders. Cell Stem Cell, 2017. 20(5): p. 659-674. e9
3. Quent, V.M.C., et al., Discrepancies between metabolic activity and DNA content as tool to assess cell proliferation in cancer research.Journal of Cellular and Molecular Medicine, 2010. 14(4): p. 1003-1013
4. Silva, L.P., et al., Measurement of DNA concentration as a normalization strategy for metabolomic data from adherent cell lines. Anal Chem, 2013. 85(20): p. 9536-42
5. Divakaruni, A.S., et al., Inhibition of the mitochondrial pyruvate carrier protects from excitotoxic neuronal death. J Cell Biol, 2017. 216(4): p. 1091-1105
6. Divakaruni, A.S., et al., Thiazolidinediones are acute, specific inhibitors of the mitochondrial pyruvate carrier. Proceedings of the National Academy of Sciences, 2013. 110(14): p. 5422-5427
7. Jiang, L., et al., Reductive carboxylation supports redox homeostasis during anchorage-independent growth.Nature, 2016. 532: p. 255
8. Choi, S.W., A.A. Gerencser, and D.G. Nicholls, Bioenergetic analysis of isolated cerebrocortical nerve terminals on a microgram scale: spare respiratory capacity and stochastic mitochondrial failure. J Neurochem, 2009. 109(4): p. 1179-91 9. Rogers, G.W., et al., High Throughput Microplate Respiratory Measurements Using Minimal Quantities Of Isolated Mitochondria. PLOS ONE, 2011. 6(7): p. e21746 10. Chan, G.K.Y., et al., A Simple High-Content Cell Cycle Assay Reveals Frequent Discrepancies between Cell Number and ATP and MTS Proliferation Assays. PLOS ONE, 2013. 8(5): p. e63583 11. Yamamoto, H., et al., Amla Enhances Mitochondrial Spare Respiratory Capacity by Increasing Mitochondrial Biogenesis and Antioxidant Systems in a Murine Skeletal Muscle Cell Line. Oxidative Medicine and Cellular Longevity, 2016. 2016: p. 11 12. Beeson, C.C., G.C. Beeson, and R.G.Schnellmann, A high throughput respirometric assay for mitochondrial biogenesis and toxicity.Anal Biochem, 2010. 404(1): p. 75-81 13. Monterisi, S., et al., PDE2A2 regulates mitochondria morphology and apoptotic cell death via local modulation of cAMP/PKA signalling. eLife, 2017.6 14. Wiley, S.E., et al., Wolfram Syndrome protein, Miner1, regulates sulphydryl redox status, the unfolded protein response, and Ca2+ homeostasis.EMBO Mol Med, 2013. 5(6): p. 904-18
声明:仅供研究使用,不用于诊断性操作。
文章来源:安捷伦细胞分析公众号
关于安捷伦细胞分析
安捷伦细胞分析平台包括xCELLigence RTCA实时细胞分析仪、Novocyte系列流式细胞仪、Seahorse能量代谢分析仪以及Synergy系列微孔板检测仪和Cytation 系列(共聚焦)细胞成像多功能微孔板检测仪。安捷伦细胞分析平台聚焦基础科研与细胞与基因治疗产品的开发全流程,作为适配新一代疗法的强大分析工具,提供多方位的细胞效力检测和深度的细胞分析,并致力于研发、生产质控以及临床的全方位检测与分析方案开发。

扫描二维码,关注安捷伦细胞分析






